
基因组DNA 中编码的遗传信息被转录为 mRNA,然后 mRNA 上的密码子在蛋白质合成过程中被转移 RNA (tRNA) 解码。tRNA 将氨基酸输送到
基因组DNA 中编码的遗传信息被转录为 mRNA,然后 mRNA 上的密码子在蛋白质合成过程中被转移 RNA (tRNA) 解码。tRNA 将氨基酸输送到核糖体,蛋白质是根据解码的遗传信息从核糖体上的氨基酸合成的。因此,tRNA 在遗传信息的翻译过程中起着关键作用。tRNA 包含许多修饰的核苷,它们调节蛋白质合成的准确性和效率。tRNA 中的修饰核苷由 tRNA 修饰酶合成。因此,揭示 tRNA 修饰酶从非底物 RNA 中选择性识别底物 tRNA 的机制;修饰酶修饰 tRNA 的时间、位置和数量,对于了解蛋白质合成机制至关重要。然而,由于缺乏识别 tRNA 修饰酶特征的高通量技术,解决这些关键问题具有挑战性。
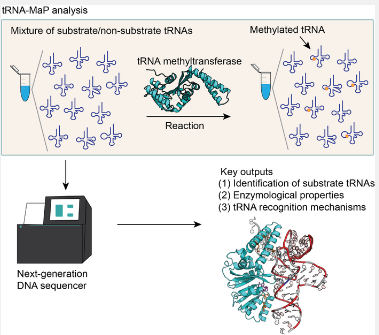
为了克服这个问题,Drs。爱媛大学的 Yamagami 和 Hori 将下一代 DNA 测序技术应用于 tRNA 修饰酶的功能分析,并开发了一种新的高通量测定方法“tRNA-MaP”。tRNA-MaP 技术可以快速筛选由 5,000 多种 RNA 组成的 RNA 池,并识别目标 tRNA 修饰酶的底物 tRNA,其灵敏度与已建立的方法相当。通过 tRNA-MaP,结合蛋白质直系学分析,我们预测了嗜热脂肪土芽孢杆菌tRNA 中的许多自然修饰。此外,我们分析了G. stearothermophilus tRNA m 1的底物识别机制A22 甲基转移酶 (TrmK) ,使用 tRNA-Map 将 tRNA 中22 位的腺苷甲基化为 1-甲基腺苷 (m 1 A22)。突变分析显示 TrmK 选择 tRNA 的一个子集作为底物。使用G. stearothermophilus tRNA Leu转录本的 240 个变体,我们发现 U8、A14、G15、G18、G19、U55、Purine57 和 A58 对于 TrmK 的甲基化很重要。此外,基于tRNA中的识别位点和TrmK的晶体结构,构建了TrmK与tRNA的对接模型。
本研究表明tRNA-Map适用于tRNA修饰酶的分析。值得注意的是,由于 tRNA-Map 可以分析来自任何生物体的任何 RNA 分子种类,甚至 DNA 分子,tRNA-Map 可用于分析除 tRNA 修饰酶之外的所有核酸相关蛋白。因此,tRNA-Map 可以加速对遗传信息流的综合理解。
声明本站所有作品图文均由用户自行上传分享,仅供网友学习交流。若您的权利被侵害,请联系我们